Africa-Press – Côte d’Ivoire. L’intelligence artificielle a été utilisée pour prédire les structures de presque toutes les protéines fabriquées par le corps humain.
Cette avancée pourrait contribuer à accélérer la découverte de nouveaux médicaments pour traiter les maladies, ainsi que d’autres applications.
Les protéines sont des éléments constitutifs essentiels des organismes vivants ; chaque cellule que nous avons en nous en est remplie.
Il est essentiel de comprendre la forme des protéines pour faire progresser la médecine, mais jusqu’à présent, seule une fraction de ces formes a été étudiée.
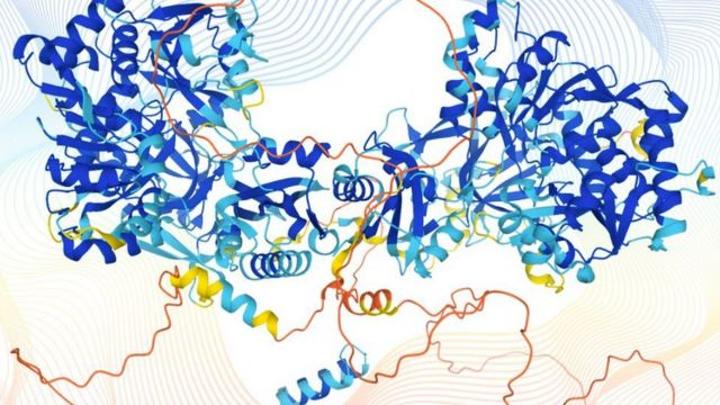
Les chercheurs ont utilisé un programme appelé AlphaFold pour déterminer la structure de 350 000 protéines appartenant à l’homme et à d’autres organismes.
Les instructions pour la fabrication des protéines humaines sont contenues dans nos génomes – l’ADN contenu dans les noyaux des cellules humaines.
On compte environ 20 000 de ces protéines exprimées par le génome humain. Collectivement, les biologistes appellent ce dispositif complet le « protéome ».
Commentant les résultats d’AlphaFold, le Dr Demis Hassabis, directeur général et cofondateur de la société d’intelligence artificielle Deep Mind, a déclaré : « Nous pensons qu’il s’agit de l’image la plus complète et la plus précise du protéome humain à ce jour.
« Nous pensons que ce travail représente la contribution la plus significative de l’IA à l’avancement des connaissances scientifiques à ce jour. « Et je pense que c’est une excellente illustration et un exemple du type de bénéfices que l’IA peut apporter à la société. » Il a poursuivi : « Nous avons hâte de voir ce que la communauté va en faire. »
Les protéines sont constituées de chaînes de blocs de construction plus petits appelés acides aminés. Ces chaînes se plient d’innombrables façons différentes, créant une forme unique en 3D. La forme d’une protéine détermine sa fonction dans le corps humain.
Les 350 000 structures protéiques prédites par AlphaFold comprennent non seulement les 20 000 structures contenues dans le protéome humain, mais aussi celles des organismes dits modèles utilisés dans la recherche scientifique, tels que E. coli, la levure, la mouche à fruits et la souris.
Ce pas de géant est décrit par les chercheurs de DeepMind et une équipe du Laboratoire européen de biologie moléculaire (EMBL) dans la prestigieuse revue Nature. AlphaFold a pu prédire avec certitude les positions structurelles de 58 % des acides aminés du protéome humain.
Les positions de 35,7 % d’entre eux ont été prédites avec un très haut degré de précision, soit le double du nombre confirmé par les expériences. Les techniques traditionnelles permettant de déterminer la structure des protéines comprennent notamment la cristallographie aux rayons X et la microscopie électronique cryogénique (Cryo-EM).
Mais aucune de ces techniques n’est facile à mettre en œuvre : « Il faut énormément d’argent et de ressources pour réaliser des structures », a confié à BBC News le professeur John McGeehan, biologiste structurel à l’université de Portsmouth.
Les formes 3D sont donc souvent déterminées dans le cadre d’investigations scientifiques ciblées, mais aucun projet n’avait jusqu’à présent déterminé de manière systématique les structures de toutes les protéines fabriquées par l’organisme.
En fait, seuls 17 % du protéome sont couverts par une structure confirmée expérimentalement. Le professeur McGeehan a expliqué les prédictions d’AlphaFold : « C’est simplement la vitesse – le fait que cela nous prenait six mois par structure et que maintenant cela prend quelques minutes. Nous n’aurions pas pu prédire que cela se produirait si vite ».
« Lorsque nous avons envoyé nos sept séquences à l’équipe de DeepMind pour la première fois, nous avions déjà les structures expérimentales pour deux d’entre elles. Nous avons donc pu les tester lorsqu’elles sont revenues. C’était un de ces moments – pour être honnête – où les poils se sont dressés sur ma nuque parce que les structures [AlphaFold] produites étaient identiques.
Le professeur Edith Heard, de l’EMBL, a souligné que : « Cette découverte sera déterminante pour notre compréhension du fonctionnement de la vie. C’est parce que les protéines représentent les blocs de construction fondamentaux à partir desquels les organismes vivants sont faits ».
« Les applications ne sont limitées que par notre compréhension ».
« Les applications que nous pouvons envisager dès à présent comprennent le développement de nouveaux médicaments et traitements pour les maladies, la conception de futures cultures capables de résister au changement climatique, et des enzymes capables de décomposer le plastique qui envahit l’environnement. Le groupe du professeur McGeehan utilise déjà les données d’AlphaFold pour aider à développer des enzymes plus rapides pour la dégradation du plastique.
Il a déclaré que le programme avait fourni des prédictions pour les protéines d’intérêt dont les structures ne pouvaient pas être déterminées expérimentalement, ce qui a permis d’accélérer leur projet de « plusieurs années ». Le Dr Ewan Birney, directeur de l’Institut européen de bioinformatique de l’EMBL, a déclaré que les structures prédites par AlphaFold constituaient « l’un des ensembles de données les plus importants depuis la cartographie du génome humain ».
DeepMind s’est associé à l’EMBL pour mettre le code AlphaFold et les prédictions de structure des protéines à la disposition de la communauté scientifique mondiale.
Le Dr Hassabis a affirmé que DeepMind prévoyait d’étendre considérablement la couverture de la base de données à presque toutes les protéines séquencées connues de la science, soit plus de 100 millions de structures.